Amazon Omics è un nuovo servizio che aiuta le organizzazioni del settore sanitario e delle scienze biologiche ad archiviare, interrogare e analizzare dati genomici, trascrittomici (per lo studio di tutte le molecole di RNA presenti in una cellula), proteomici (per lo studio su grande scala delle proteine contenute in una cellula in un dato momento) e altri dati omici e quindi generare insight da tali dati per migliorare la salute e far progredire le scoperte scientifiche. Omics è progettato per supportare analisi su larga scala e ricerca collaborativa in modo da poter archiviare e, insieme ad altri servizi AWS, analizzare i dati del genoma per intere popolazioni. Amazon Omics automatizza inoltre il provisioning e il dimensionamento dei flussi di lavoro bioinformatici, in modo da poter eseguire pipeline di analisi su scala di produzione e dedicare più tempo alla ricerca e all’innovazione.
Con Amazon Omics è possibile combinare dati genomici, biologici e sulla salute della popolazione per generare informazioni dettagliate e offrire un’assistenza più personalizzata con l’analisi multimodale. Ad esempio, si possono addestrare modelli ML con Amazon SageMaker per aiutare i ricercatori a prevedere se gli individui sono predisposti a determinate malattie. O combinare i dati del genoma di un individuo con la sua storia medica da Amazon HealthLake per fornire diagnosi migliori e piani di trattamento personalizzati.
Amazon Omics consente inoltre di importare e combinare i dati con altri set di dati di riferimento disponibili pubblicamente nel Registry of Open Data di AWS, come il 1000 Genomes Project, che può essere utilizzato come controllo per comprendere il rischio di malattia; il Genome Aggregation Database (gnomAD), che consente di inserire le frequenze alleliche della popolazione per aprire la porta all’individuazione delle malattie, e oltre 60 altri set di dati genomici.
Auditor di terze parti valutano la sicurezza e la conformità di Amazon Omics nell’ambito di diversi programmi di conformità AWS. Questi includono HIPAA, FedRAMP, GDPR e altri. Amazon Omics è ora disponibile nelle region europee Irlanda, Londra e Francoforte, oltre che in region degli Stati Uniti e in Asia.
Direttamente nella console Omics è possibile importare e normalizzare petabyte di dati in formati ottimizzati per l’analisi. Amazon Omics fornisce flussi di lavoro scalabili e strumenti integrati per la preparazione e l’analisi dei dati omici ed effettua automaticamente il provisioning e il dimensionamento dell’infrastruttura cloud sottostante. Quindi, ci si può concentrare sul progresso della scienza e tradurre le scoperte in diagnostica e terapie.
Amazon Omics è stato annunciato da Adam Selipsky, Ceo di Amazon Web Services nel corso del keynote tenuto il secondo giorno di AWS re:Invent 22
I componenti di Amazon Omics
Amazon Omics ha tre componenti principali:
- Il primo componente è lo storage di oggetti compatibile con Omic per archiviare, scoprire e condividere i dati di sequenza grezzi in modo efficiente, sicuro e a basso costo. Il secondo componente è Omics Workflows, che consente di eseguire flussi di lavoro bioinformatici riproducibili per elaborare i dati di sequenza grezzi su larga scala sia in Omics Storage sia in S3, eliminando tutto il lavoro pesante indifferenziato associato all’esecuzione di questi flussi di lavoro. E il terzo componente è Omics Analytics, che semplifica l’analisi attraverso varianti (o mutazioni) e annotazioni pronte per le query. Mentre questi componenti saranno spesso utilizzati insieme, è possibile anche sfruttarli in modo autonomo.

Archiviazione dei dati Omics
L’archiviazione dei dati Omics consente di archiviare e condividere petabyte di dati omici in modo efficiente. È possibile creare datastore e importare dati di esempio nella console Omics o svolgere lo stesso lavoro nell’interfaccia a riga di comando di AWS (AWS CLI).
Amazon Omics offre opzioni di storage compatibile con metodi omici per i dati di riferimento e di sequenza, in grado di ridurre il TCO (Total Cost of Ownership) per l’archiviazione di dati di sequenza grezzi (ad esempio BAMs, CRAMs, FASTQs). Facilita inoltre la condivisione e la governance dei dati attraverso una combinazione di controllo degli accessi basato su attributi e metadati ricercabili specifici del dominio. Amazon Omics rende disponibili due classi di storage: active, per i dati facilmente accessibili, e archive, per l’archiviazione a basso costo e a lungo termine dei dati omici. L’archiviazione automatica è attiva per impostazione predefinita, il che significa che Amazon Omics sposterà automaticamente i dati nella classe di storage più economica se non vi si accede regolarmente (per più di 30 giorni), in modo simile alla classe di storage Intelligent–Tiering di Amazon Simple Storage Service (Amazon S3), con conseguenti risparmi sui costi. È importante sottolineare che, poiché tutti gli strumenti di sequenziamento sono classificati in gigabasi per esecuzione (ad esempio, un genoma umano contiene tre miliardi di basi o tre gigabase), in cui i clienti in genere progettano i loro panel clinici in gigabasi per test, lo storage Amazon Omics è progettato per essere specifico del dominio per numero di gigabasi acquisite. Ciò garantisce un’archiviazione ottimale dei dati di sequenziamento grezzi su larga scala, che possono raggiungere centinaia di migliaia di genomi all’anno. Inoltre, offre ai clienti la flessibilità di scegliere qualsiasi piattaforma o tecnologia di sequenziamento (ad esempio, letture brevi o letture lunghe) e la prevedibilità dei prezzi senza preoccuparsi della variabilità dei costi di storage tra le piattaforme di sequenziamento.
Analytics Transformations
L’output dell’analisi del sequenziamento del DNA sono varianti genomiche grezze sotto forma di Variant Call Files (VCF) e Genomic Variant Call Files (gVCF). I ricercatori vogliono interrogare queste varianti e identificarne il significato clinico assegnando loro un significato attraverso annotazioni (ad esempio, identificando una variante che si traduce in un gene difettoso che produce una proteina cancerogena). Questi VCF basati su testo, tuttavia, non sono ottimizzati per l’esecuzione di query data la loro natura semistrutturata. Mentre i genomi possono essere analizzati a livello individuale, molti ricercatori stanno cercando di interrogare migliaia di varianti su molti geni contemporaneamente per capire come la variazione genomica, unita ai corrispondenti dati clinici, possa influenzare la salute umana o prevedere i risultati clinici.
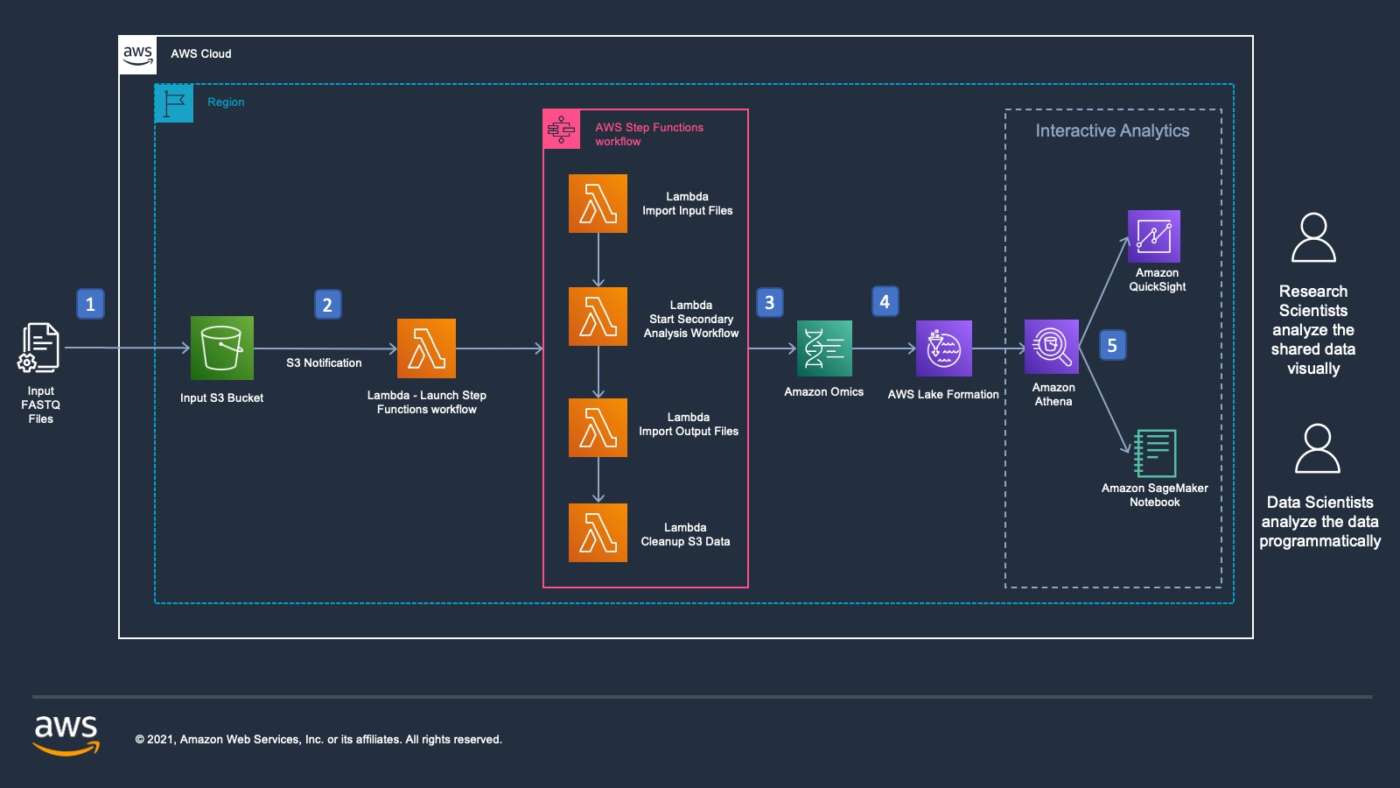
Amazon Omics permette di importare il proprio VCF in un Variant Store e trasformarlo in uno schema pronto per le query disponibile come Apache Iceberg Table; supporta inoltre l’importazione di annotazioni varianti in un archivio annotazioni. È possibile governare l’accesso tramite AWS Lake Formation e applicare un controllo granulare degli accessi per filtrare i singoli pazienti. Questo aiuta a definire coorti di pazienti personalizzate e gestire il consenso dei pazienti per i regimi di conformità, come il GDPR, senza dover copiare i dati. Ciò consente inoltre di eseguire query e analisi di queste varianti utilizzando Amazon Athena e di unire dati provenienti da altre modalità, come i dati clinici in Amazon HealthLake o nel catalogo dati AWS Glue. Amazon QuickSight può essere utilizzato come interfaccia visiva per definire una coorte di pazienti in base a vari attributi, come una particolare mutazione o osservazioni cliniche in Amazon HealthLake. Amazon SageMaker può essere utilizzato per creare e distribuire molti modelli di machine learning su questa coorte, in modo rapido ed efficiente, per previsioni basate sull’intelligenza artificiale come il rischio di malattia di un paziente o l’efficacia complessiva di un particolare farmaco.
Flussi di lavoro bioinformatici
Amazon Omics fornisce risorse di elaborazione gestite per eseguire flussi di lavoro bioinformatici riproducibili, che contengono script di una serie di attività coordinate per distillare grandi quantità di dati di sequenza grezzi, dallo storage Amazon Omics o Amazon S3, a piccole quantità di dati analitici (come mutazioni del genoma, note come varianti, o conteggi di espressione genica). A sua volta, questo rimuove tutto il lavoro pesante indifferenziato associato all’esecuzione e alla gestione di questi flussi di lavoro su larga scala.
È possibile eseguire script scritti in una varietà di linguaggi di flusso di lavoro bioinformatici, come Nextflow o WDL (Workflow Description Language) e immagini Docker corrispondenti. I clienti specificano quindi le risorse di calcolo (vCPU e memoria) necessarie per ogni attività. Amazon Omics gestisce la pianificazione e il provisioning delle risorse per eseguire il flusso di lavoro, gestisce i tentativi ed effettua il provisioning dei file system condivisi.
Amazon Omics consente di definire gruppi di esecuzione per limitare il numero massimo di esecuzioni simultanee per un flusso di lavoro e un utente specifici, consentendo di gestire i costi e monitorare più progetti. I parametri e i log di esecuzione sono accessibili tramite Amazon Cloud Watch o la console Amazon Mics. Amazon Omics consente inoltre di monitorare la provenienza e la derivazione dei dati tenendo traccia del flusso di lavoro eseguito per un determinato input e degli output generati da tale esecuzione.


