Comprendere come funziona la memoria umana non è un esercizio teorico. È uno dei problemi scientifici più complessi e concreti della medicina moderna, perché da lì passano la comprensione di patologie come Alzheimer, demenze e disturbi cognitivi. Al Marine Biological Laboratory di Woods Hole, negli Stati Uniti, un gruppo di ricercatori sta affrontando questa sfida usando un approccio radicalmente diverso dal passato: combinare intelligenza artificiale, realtà virtuale immersiva e calcolo accelerato su GPU.
L’obiettivo non è simulare il cervello, ma osservarlo davvero, a una scala e con un livello di dettaglio che fino a pochi anni fa erano semplicemente impraticabili. Il progetto nasce dalla necessità di analizzare quantità di dati biologici enormi, tridimensionali e altamente strutturati, che superano di gran lunga le capacità degli strumenti tradizionali di microscopia e analisi.
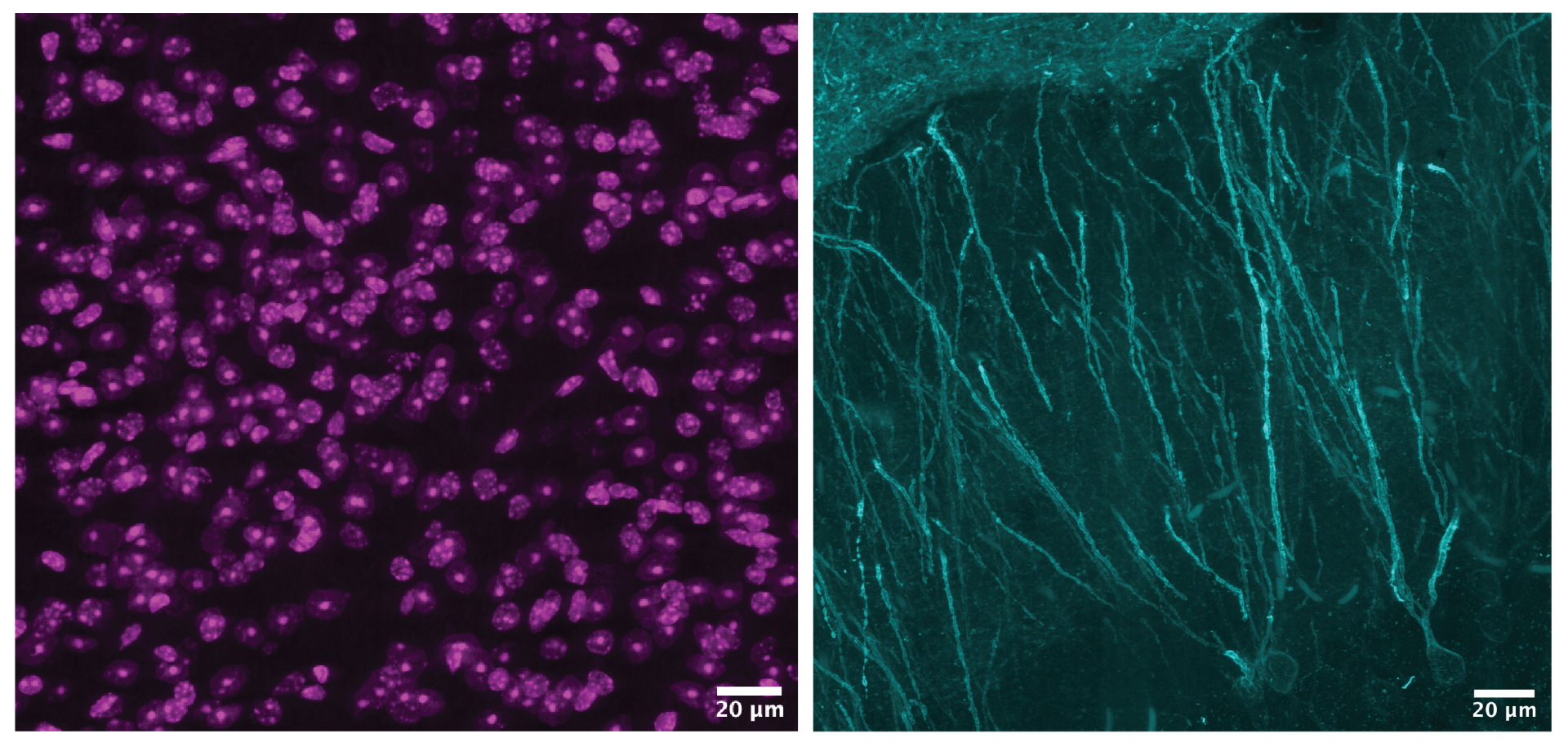
L’ippocampo come foresta di neuroni
Il lavoro si concentra sull’ippocampo, una struttura cerebrale cruciale per la formazione e il recupero dei ricordi. Dal punto di vista anatomico, l’ippocampo può essere immaginato come una foresta estremamente densa: ogni neurone è un tronco che si ramifica in migliaia di dendriti, su cui si distribuiscono proteine fondamentali per la memoria.
Il problema è che le proteine di interesse rappresentano una frazione minima, circa l’1%, di tutte quelle presenti nel tessuto. Identificarle equivale a cercare pattern specifici in un volume tridimensionale composto da miliardi di elementi. Farlo in due dimensioni, con immagini piatte o sezioni isolate, significa perdere contesto, relazioni spaziali e continuità strutturale.
Qui sta il collo di bottiglia storico della neuroscienza: i dati esistono, ma non sono leggibili dall’essere umano senza un nuovo tipo di interfaccia.
Quando il limite non è la scienza, ma la visualizzazione
Il progetto del MBL parte da una constatazione semplice e brutale: l’analisi manuale di questi dataset è impossibile, mentre l’automazione pura rischia di perdere segnali biologicamente rilevanti. La soluzione adottata non è sostituire l’uomo con l’AI, ma potenziare la percezione umana.
Per farlo, il laboratorio ha integrato workstation ad alte prestazioni con GPU NVIDIA RTX, capaci di gestire rendering volumetrici 3D estremamente complessi in tempo reale. Non si parla di gigabyte, ma di decine di terabyte di dati volumetrici, acquisiti tramite tecniche avanzate di imaging biologico.
Senza accelerazione GPU, questi volumi richiederebbero tempi di elaborazione incompatibili con la ricerca. Con RTX, diventano esplorabili in modo interattivo.
Realtà virtuale come strumento scientifico
La vera svolta arriva con l’uso della realtà virtuale immersiva. Grazie al software syGlass, i ricercatori possono letteralmente entrare all’interno del tessuto cerebrale digitalizzato. Non osservano più immagini su uno schermo: camminano dentro il dataset, ruotano le strutture, isolano proteine, confrontano regioni e riconoscono pattern tridimensionali che sarebbero invisibili su un piano bidimensionale.
Questo approccio sfrutta una capacità tipicamente umana: il riconoscimento visivo di forme e relazioni spaziali complesse. La VR non è un gadget, ma una interfaccia cognitiva che consente al cervello umano di lavorare su dati biologici ad altissima densità informativa.
AI e GPU: accelerazione, non sostituzione
L’intelligenza artificiale entra in gioco come supporto all’analisi, non come arbitro finale. Algoritmi di machine learning aiutano a segmentare le immagini, evidenziare aree di interesse e suggerire correlazioni, ma la validazione resta umana.
Le GPU RTX svolgono un ruolo chiave anche qui: l’addestramento e l’esecuzione dei modelli AI avvengono localmente, in tempi compatibili con l’interazione in VR. Questo consente un ciclo continuo di osservazione, ipotesi e verifica, senza dover esternalizzare il calcolo o attendere elaborazioni offline.
Dal laboratorio alla formazione
Uno degli aspetti più interessanti del progetto è l’impatto sulla formazione scientifica. L’infrastruttura sviluppata al MBL ha permesso di coinvolgere anche studenti non ancora universitari in attività di ricerca reale. Indossando un visore VR, gli studenti hanno potuto esplorare dataset autentici e contribuire all’identificazione di strutture biologiche rilevanti.
Non è divulgazione, è ricerca assistita, resa possibile da un’interfaccia che abbassa drasticamente la barriera di accesso a dati estremamente complessi.
Implicazioni cliniche e scientifiche
Capire come le proteine si distribuiscono e interagiscono nell’ippocampo significa aprire nuove strade per la diagnosi precoce delle malattie neurodegenerative. Alterazioni microscopiche nella posizione o nella densità di alcune proteine possono essere i primi segnali di un processo patologico.
La possibilità di osservare questi fenomeni in 3D, nel loro contesto strutturale reale, rappresenta un cambio di paradigma rispetto all’analisi frammentata del passato.
Oltre il mito del “GPU per videogiochi”
Questo progetto smonta definitivamente l’idea che le GPU siano strumenti specialistici confinati al gaming o alla grafica. In questo contesto, le GPU RTX diventano strumenti scientifici di base, senza i quali la ricerca semplicemente non può avanzare.
Non accelerano solo il calcolo: accelerano la comprensione. Consentono di trasformare dataset ingestibili in ambienti esplorabili, e di restituire all’essere umano un ruolo centrale nell’interpretazione dei dati.
Una nuova forma di osservazione scientifica
Il lavoro del Marine Biological Laboratory dimostra che la frontiera della scienza non passa solo da nuovi modelli teorici, ma da nuovi modi di vedere. AI, realtà virtuale e GPU non sono tecnologie accessorie: sono gli strumenti che rendono osservabile ciò che prima era invisibile.
La memoria umana resta uno dei grandi misteri della biologia, ma oggi, grazie a queste tecnologie, non è più un territorio completamente opaco. È un ambiente che possiamo esplorare, analizzare e, passo dopo passo, comprendere.